THREAD—L’ARN
Avec la technologie des vaccins à ARN messager, le monde a ‘découvert’ l’ARN. Et beaucoup l’ont associé à des biotechnologies. Mais l’ARN, c’est AVANT TOUT un type de macromolécules universelles sur Terre et essentielles à toutes les cellules.⬇️⬇️⬇️
[25 tweets] https://t.co/knKy9LAZtV
[1/25] ARN, ça signifie « acide ribonucléique ». Il a été découvert en 1869 en même temps que l’ADN et par la même personne (F. Miescher) [1,2]. Mais la caractérisation chimique de l’ARN s’est étalée sur 84 ans (1869-1953) [3]. Je t’en ai déjà parlé ici.
https://t.co/akB1g3gtt9
[2/25] Avant de commencer, faut que tu saches que l’ARN, c’est un type de macromolécule qu’on trouve chez TOUS les êtres vivants. Absolument tous. Il n’y a aucune exception. Et on en trouve même beaucoup, aussi bien en termes de quantité que de types différents.
[3/25] Je peux citer [4-9] les
➡️ARN messagers (ARNm)
➡️ARN de transfert (ARNt)
➡️ARN ribosomiques (ARNr)
➡️microARN
➡️piARN
➡️petits ARN nucléaires (snARN)
➡️hnARN et les snoARN
➡️ARN 6S (ou 4.5S)
➡️lncARN
➡️ARN Y
➡️ARN vault
➡️ARN circulaires
Etc…
[4/25] D’abord, l’ARN, c’est la forme sous laquelle TOUS les gènes SANS EXCEPTION sont « exprimés ». D’ailleurs, c l’une des définitions « moléculaires » d’un gène: une région de l’ADN qui est transcrite sous la forme d’un ARN [10,11].
[5/25] Le fait que les ARN soient le résultat de l’expression d’un gène n’a commencé à être découvert qu’en 1961 par Jacques Monod, François Jacob et Sydney Brenner, avec la découverte de l’ARN messager —la première forme d’expression des gènes de protéines [12].
[6/25] TOUS les ARN sont des chaînes de briques plus petites qu’on appelle des ribonucléotides, et qui sont des cousins des désoxyribonucléotides qui constituent l’ADN. Il y a 4 ribonucléotides de base qu’on abrège A, U, G et C⬇️. https://t.co/P0X7e2lboL

[7/25] Tous les ARN sont des assemblages linéaires (des polymères) de ces ribonucléotides et le nombre de ces ribonucléotides et l’ordre spécifique dans lequel ils sont assemblés dans un brin d’ARN, on appelle ça sa séquence⬇️. https://t.co/dkNtPItNt0

[8/25] Les ribonucléotides sont certes des « cousins » des désoxyribonucléotides de l’ADN, mais il y a quand même des différences⬇️ —elles ont l’air minimes, mais en réalité elles changent drastiquement les propriétés de l’ARN vis-à-vis de celles de l’ADN. https://t.co/rUoEUPYPBj

[9/25] D’ailleurs, si l’ARN est la forme d’expression de tous les gènes, pourquoi personne n’avait-il découvert ça AVANT 1961? Eh bien ça tient à l’une des propriétés majeures de tous les ARN: leur fragilité.
[10/25] La présence de l’hydroxyle (OH) sur le ribose —qui est absent du désoxyribose de l’ADN— est problématique. Ce dernier peut réagir (chimiquement parlant) sur la liaison phosphodiester à côté de lui et la cliver. On appelle ça une hydrolyse⬇️. https://t.co/tO84UfHWuY

[11/25] Mais attention, cette réaction se produit invariablement sur TOUS les ARN s’ils sont dans l’eau et que le pH est neutre (et c’est alors très lent) ou basique (c’est-à-dire supérieur à 7 et là, et là c’est rapide).
[12/25] L’ARN se dégrade tout seul s’il est dans un milieu aqueux (avec de l’eau): pas facile de l’extraire et de l’étudier. Ensuite, tous les organismes sécrètent des enzymes appelées RNases [13,14] qui… hydrolysent l’ARN⬇️. C’est l’une des lignes de défense contre les virus. https://t.co/OZUeNRokAu

[13/25] Bref, dans les cellules, l’ARN, faut qu’il soit protégé de l’hydrolyse spontanée dans l’eau, mais aussi de la dégradation par les nombreuses ribonucléases (RNases). Et dès que tu les détruits pour étudier ce qu’elles contiennent… il se dégrade vite. Très vite !
[14/25] Aujourd’hui, on sait gérer ça. Y a des techniques pour éviter ou inhiber les RNases et maintenir un pH favorable pour réussir à préserver l’ARN in vitro, mais à l’époque, c’était pas le cas. D’où le fait que son rôle central nous a échappé assez longtemps.
[15/25] Bref. Chaque ARN est donc un brin qui possède une séquence spécifique de ribonucléotides et, de cette séquence, dépend le REPLIEMENT en 3D qu’il peut adopter. Oui, comme pour les protéines, sauf que là, t’as des ribonucléotides.
[16/25] Pour l’ADN, t’avais deux brins complémentaires formant une double-hélice régulière grâce à la complémentarité d’interaction entre les bases⬇️. L’ARN est constitué en général d’UN SEUL brin et il n’y a pas de 2e brin pour contraindre sa linéarité.
https://t.co/fsxHJMQVyX
[17/25] Du coup, il peut se REPLIER SUR LUI-MÊME en fonction des paires de bases qu’il peut former avec ses propres nucléotides. Dans l’ADN, les paires, c’était entre A et T et entre C et G. Dans l’ARN, t’as pas de T, mais un U qui a les mêmes propriétés de complémentarité.
[18/25] En conséquence, tu peux faire des paires G—C (comme dans l’ADN) mais aussi des paires A—U. Mais contrairement à l’ADN, là, tu peux aussi avoir des paires de bases G—U en plus. Y en a d’autres, mais ce sont les 3 principales⬇️. https://t.co/lE059bp6rL

[19/25] Je te donne l’exemple d’un ARN de transfert, une chaîne de 76 nucléotides qui se replie sur elle-même selon ces interactions⬇️. Et tu formes de structures dites « secondaires » en tiges, en boucles, etc. https://t.co/ve18CnF9ol

[20/25] Ensuite, ces structures secondaires interagissent entre elles et forment une structure tridimensionnelle caractéristique, le REPLIEMENT de l’ARN. Là⬇️, je te mets l’ARNt en 3D. Oui, c’est le même principe que pour les chaînes d’acides aminés dans les protéines. https://t.co/BmY1hAnz0a
[Film] Et ici⬇️, je te mets encore une animation avec un ARNt replié en 3D, mais cette fois avec une représentation simplifiée (l’équivalent des rubans dont je t’avais parlé pour les protéines). https://t.co/lcdtIuG2iL
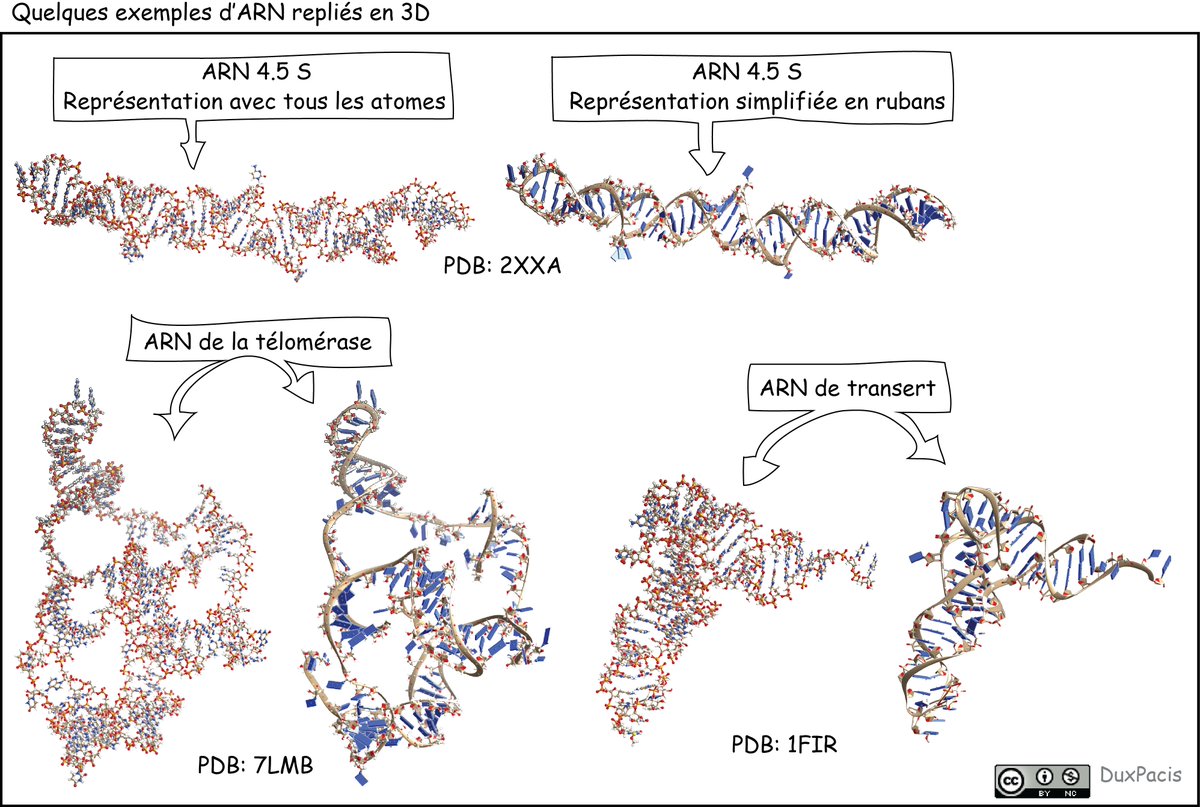
[21/25] Voilà des exemples de plusieurs ARN⬇️, qui se replient en 3D grâce aux paires de bases qu’ils peuvent former entre différentes régions de la chaîne qui les constitue. https://t.co/yW1ooIHfLI

[22/25] Dans l’ADN, t’avais 4 désoxyribonucléotides différents (A, T, G, C) et dans l’ARN t’en a 4 aussi (A, U, G, C). Mais tu sais quoi? Une fois qu’un ARN est synthétisé, t’as des tas de modifications qui peuvent avoir lieu sur les nucléotides [14-18].
[23/25] Et il existe des centaines de nucléotides modifiés: modification de la base azotée (uracile en pseudouridine, par ex.), modification des hydroxyles du ribose (méthylations), et bien d’autres encore.
[24/25] Par exemple, dans l’ARN de transfert d’avant⬇️, en réalité, t’as des dizaines de modifications de ce type. T’as la pseudouridine (lettre gecque psi) qui est très présente, mais y en a d’autres aussi (partout où le nucléotide est annoté avec d’autres lettres) [19]. https://t.co/EzzFIDgwVs

[25/25] Ces modifications, tu les trouves dans les ARN de transfert, les ARN ribosomiques et même dans les ARN messagers⬇️, en particulier la méthylation (ajout d’un CH3) sur la position 2’ du ribose (qui évite la sensibilité à l’hydrolyse dans les cellules !) et la pseudouridine https://t.co/r53Rt9oMyT

[FIN] Bref, tu comprends que les ARN c’est beaucoup plus complexe que des chaînes de seulement 4 lettres (A, U, G, C): ils peuvent être modifiés et ils se replient en trois dimensions en molécules complexes. Maintenant que tu sais tout ça, on pourra passer au niveau supérieur...
...un de ces jours. Mais en attendant, bah je te dis merci d’avoir lu jusque-là, et je te dis à bientôt !
Les références, comme d'habitude, sont en-dessous
[REFERENCES]
[1] Dahm, R. (2008) Discovering DNA : Friedrich Miescher and the early years of nucleic acids research, Hum Genet, 122, 565-581.
[2] Thess, A, Hoerr, I, et al. (2021) , Biol Chem, 402(10), 1179-1185
[3] Frixione, E, Ruiz-Zamarripa, L (2019) The "scientific catastrophe" in nucleic acids , J Biol Chem, 15;294(7):2249-2255
[4] Zhang P, Wu W, Chen Q, Chen M. Non-Coding RNAs and their Integrated Networks. J Integr Bioinform. 2019 Jul 13;16(3):20190027.
[5] ARN en general: Burgess D. A comprehensive view of human RNAs. Nat Rev Genet. 2021 ;22(8):481. doi: 10.1038/s41576-021-00393-1.
[6] Les ARNt: Berg MD, Brandl CJ. Transfer RNAs: diversity in form and function. RNA Biol. 2021 ;18(3):316-339. doi: 10.1080/15476286.2020.1809197
[7] Su Z, Wilson B, Kumar P, Dutta A. Noncanonical Roles of tRNAs: tRNA Fragments and Beyond. Annu Rev Genet. 2020 ;54:47-69.
[8] Les snoARN: Lui L, Lowe T. Small nucleolar RNAs and RNA-guided post-transcriptional modification. Essays Biochem. 2013;54:53-77.
[9] He C, Wang K, et al. Roles of Noncoding RNA in Reproduction. Front Genet. 2021 ;12:777510.
[9] Hahne JC, Lampis A, Valeri N. Vault RNAs: hidden gems in RNA and protein regulation. Cell Mol Life Sci. 2021 Feb;78(4):1487-1499. doi: 10.1007/s00018-020-03675-9.
[10] Le Guyader H Qu'est-ce qu'un gène? Le Courrier de l'environnement de l'INRA, Paris: Inst. nat. de la recherche agro. Délég. permanente à l'environnement, 2001, pp53-63.
[11] Portin, P., and A. Wilkins. 2017. The Evolving Definition of the Term "Gene"', Genetics, 205: 1353-64
[12] Cobb M. Who discovered messenger RNA? Curr Biol. 2015 Jun 29;25(13):R526-32.
[13] Weick EM, et al. Helicase-Dependent RNA Decay Illuminated by a Cryo-EM Structure of a Human Nuclear RNA Exosome-MTR4 Complex. Cell. 2018 Jun 14;173(7):1663-1677.e21.
[14] Ogami K, Suzuki HI. Nuclear RNA Exosome and Pervasive Transcription. Int J Mol Sci. 2021 ;22(24):13401.
[15] Accornero F, et al. From canonical to modified nucleotides: balancing translation and metabolism. Crit Rev Biochem Mol Biol. 2020 ;55(6):525-540.
[16] Boo SH, Kim YK. The emerging role of RNA modifications in the regulation of mRNA stability. Exp Mol Med. 2020 ;52(3):400-408.
[17] Lin TY, Mehta R, Glatt S. Pseudouridines in RNAs: switching atoms means shifting paradigms. FEBS Lett. 2021 Sep;595(18):2310-2322.
[18] Shigi N. Biosynthesis and Degradation of Sulfur Modifications in tRNAs. Int J Mol Sci. 2021 Nov 3;22(21):11937.
[19] Vendeix FA, et al. Human tRNA(Lys3)(UUU) is pre-structured by natural modifications f. J Mol Biol. 2012 Mar 2;416(4):467-85.
